
news
新闻中心
FISH检测ROS1重排为何一绿一融合是阳性?
背景介绍
c-ros肉瘤致癌因子-受体酪氨酸激酶(ROS proto-oncogene 1 , receptor tyrosine kinase, ROS1) 基因定位于6q22.1,编码一种受体酪氨酸激酶(RTK),与细胞的生长、增殖、分化和生存有关。在NSCLC中ROS1基因主要与CD74、EZR、SLC34A2和GOPC等14种基因发生融合,并持续激活ROS1酪氨酸激酶区及下游信号通路,进而引起肿瘤的发生发展。
信号模式
GOPC基因位于6q22.1区域,与ROS1基因相隔200kb。GOPC基因与ROS1基因融合时,会产生ROS1与GOPC中间区域内的片段缺失,就会在FISH检测中看到一绿一融合的信号模式。
EZR基因位于6q25.3区域,与ROS1基因相隔4Mb。ERZ基因与ROS1基因融合时,会产生染色体内倒位,形成典型的一绿一红一融合信号模式。
当ROS1与其他基因发生重排时,ROS1基因5’端转移至其他染色体区域,形成了典型的一绿一红一融合信号模式。
根据现有探针设计方案,笔者发现不同探针设计方案可能在检测ROS1基因重排时并不能检测出非典型的一绿一融合信号模式。
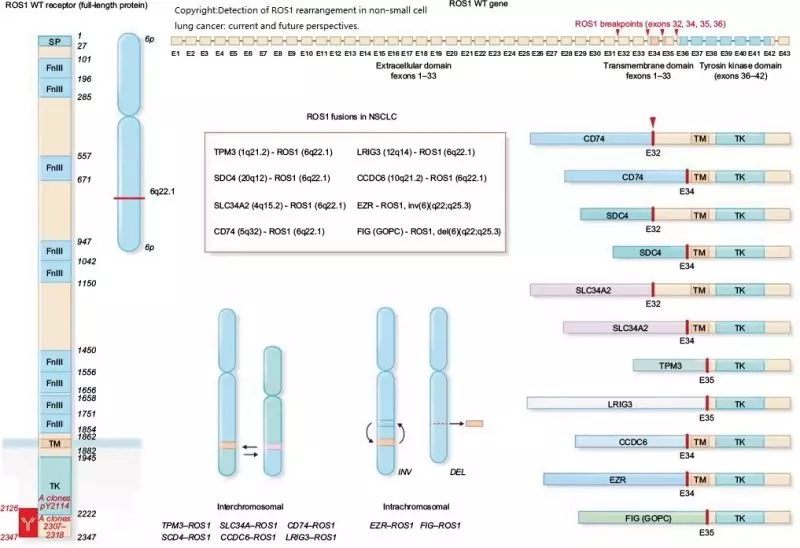
图1 ROS1基因重排模式
探针设计
不同公司的探针,其设计方案可能有所不同,下面列举市面上主流四家公司ROS1基因断裂探针的设计方案,比较各探针设计的区别。
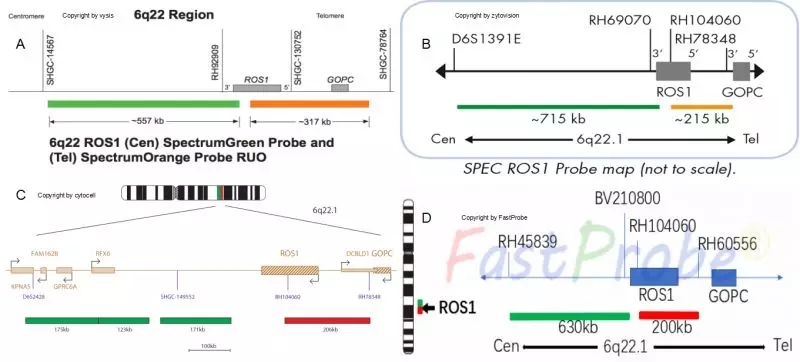
图2 ROS1断裂探针设计
图2中可以看到,A公司ROS1基因断裂探针设计方案采用绿色染料标记ROS1基因3’端区域(557kb),橘红色染料标记ROS1基因5’端区域(317kb),5’端长度317kb覆盖了GOPC基因。BCD公司ROS1基因断裂探针5’端区域长约200kb,并未覆盖GOPC基因。
比较两种设计方案,A公司橘红色染料标记ROS1基因5’端区域包括ROS1基因和GOPC基因,当发生GOPC-ROS1重排而导致GOPC与ROS1中间区域缺失时,由于该设计会残留100kb左右片段,形成在显微镜下可见的红色信号点,仍然形成一个融合信号,所以该设计方案无法检测ROS1和 GOPC是否发生重排,存在漏检风险。
BCD公司橘红色染料标记ROS1基因5’端区域不包括GOPC基因,当ROS1和 GOPC发生重排,形成GOPC-ROS1融合基因时,引起两个基因中间区域200kb左右红色区域缺失,导致单绿(1G1F)阳性信号,与《ROS1阳性非小细胞肺癌诊断病理专家共识》中ROS1结果判读一致。
从ROS1基因断裂探针设计可以看出,不同厂家的设计方案会有差别,该差别会导致临床漏检,存在较大的风险。当我们在选购不同探针时,需要认真研究探针设计方案,尽量减少临床漏检和错检情况的出现。